PERGUNTAS DE BIOLOGIA MOLECULAR
GRUPO 1
Pedro Henrique Aguiar
P:Defina "Origem de Replicação".
R:Origem de Replicação (ou Ori) é uma região na fita de DNA
que possui características incomuns, como sequências de
nucleotídeos repetidos, que são reconhecidas por um complexo
enzimático e atuam como sítio de ligação para essas enzimas
iniciando o processo de replicação do DNA. Uma molécula de
DNA tem várias origens de replicação, algumas são escolhidas
com mais frequência.
Robson Henrique
Sobre o modelo do DNA de Watson-Crick, é correto afirmar que:
A. O esqueleto de cada fita do DNA é uma cadeia polimérica de açúcar-fosfato.
B. As fitas do DNA são anti-paralelas, e se enrolam em espiral em torno do eixo da hélice em sentidos opostos.
C. As sequüências de bases nas duas fitas são determinadas pela formação de pontes de hidrogênio entre adenina e timina ou entre guanina e citosina.
D. O açúcar presente nas cadeias é a desoxirribose.
E. Todas as alternativas acima são características da estrutura do DNA segundo o modelo de Watson-Crick.
Gina Mantilla
P:Sobre vetores de clonagem responda: quando é recomendável o uso de plasmídeos, fagos e cosmídeos na clonagem de um inserto que codifica um gene de interesse?
R: Plasmídeos: é usado em insertos pequenos e que possuem pouca especificidade para se ligarem ao vetor.
Fagos: Para clonar fragmentos muito grandes.
Cosmídeos: Parecido com o fago, porém é mais fácil para manipular por não possuir especificidade.
Paula Coelho
P: Os primeiros seres vivos possuíam RNA como molécula responsável por armazenar informação genética. Todos os organismos vivos estudados apresentam seu material genético composto por DNA. Discuta o processo de substituição do mundo RNA para o mundo DNA ao longo da evolução.
R: A molécula de DNA por ser mais estável que a molécula de RNA é mais eficiente em armazenar informação. Muito da instabilidade do RNA deve-se à reação espontânea de desaminação da citosina gerando uma uracila. Essa uracila, por sua vez, se não for reparada, pode levar a uma transição C→T. Como a molécula de DNA não possui uracila, quando suas citosinas desaminam e geram uracilas, essas são rapidamente reconhecidas e removidas pela enzima uracila DNA-glicosilase. Essa reação resulta na formação de sítios abásicos que são subseqüentemente preenchidos.
João Pedro Vieira
P: Descreva em linhas gerais o procedimento de purificação de DNA plasmidial pela técnica de Mini-prep (técnica de lise alcalina)
R: Centrifugação da cultura→ Ressuspensão em tampão→adição de NAOH/SOS: lise bacteriana em pH alcalino→ neutralização do lisado (com acetato de potássio)→ centrifugação e descarte do DNA cromossômico→ DNA plasmidial no sobrenadante (com adição de isopropanol).
GRUPO 2
Questão 1: Ellen Christie Pereira
Hoje em dia, a maior parte da insulina consumida por milhões de diabéticos é produzida por bactérias. Sabendo que o genoma humano contém íntrons e que as bactérias não são capazes de remove-los, responda: que processo é feito para que seja possível a obtenção de insulina via bactéria?
Resposta:
A partir do RNAm da pró-insulina, extraído de um pâncreas humano, obtém-se um DNAc da pró-insulina. Isso é feito, utilizando-se a enzima transcriptase reversa, formando um híbrido de DNA-RNA. É feito, então, um grampo com a própria extremidade 3’OH da fita de DNA já formada, servindo com primer para a síntese do filamento complementar de DNA. A alça é removida por digestão.
A pró-insulina é precursora das cadeias alfa e beta da insulina. Cada uma dessas partes é inserida em um plasmídeo que infectam duas bactérias distintas. Isso porque uma bactéria apenas não é capaz de sintetizar as duas cadeias. Um clone produzirá a cadeia alfa e o outro a cadeia beta. Essas unidades serão purificadas e unidas in vitro com ponte de enxofre.
Questão 2: Júlia Silveira Fahel
A técnica de clonagem é útil na construção de bibliotecas genômicas, expressão de proteínas etc. Implica no uso de um vetor onde é inserido um fragmento de DNA em estudo. Um dos vetores de clonagem mais comumente utilizados são os plasmídeos. Alguns deles foram geneticamente modificados para otimizar a inserção de moléculas de DNA e para facilitar a seleção de colônias bacterianas que contém o plasmídeo com o fragmento de DNA desejado.
Cite e dê a função dos principais componentes de um plasmídeo geneticamente modificado e explique porque a cor branca das bactérias transformadas indica que elas contém o plasmídeo com o fragmento de DNA inserido.
Resposta:
Um plasmídeo geneticamente modificado, em geral, contém um ou mais genes de resistência a drogas cuja função é selecionar as bactérias transformadas pelo plasmídeo. Contém também um sítio de clonagem múltipla que apresenta muitos sítios-alvo de enzimas de restrição. É neste sítio que são inseridos os fragmentos de DNA. Alguns plasmídeos contém ainda o gene Lac Z responsável pela produção da enzima b -galactosidase. Esse gene facilita a seleção das bactérias transformadas.
O gene Lac Z está em contiguidade com o sítio de múltipla clonagem. Quando no plasmídeo é inserido um fragmento de DNA, esse gene é interrompido e não há produção da b -galactosidase. Essa enzima age sobre um substrato incolor chamado X-gal que é adicionado ao meio convertendo-o em um corante azul. A colônia que apresenta o plasmídeo sem o gene Lac Z interrompido fica azul. A colônia que apresenta esse gene interrompido torna-se branca e é selecionada.
Questão 3:Mariana Amorim
Para seqüenciar fragmentos de DNA, atualmente, usa-se o seqüenciamento automático com didesoxi-NTP marcados com fluorescência.
Os ddNTP não possuem OH na posição 3’ da desoxirribose, mas apenas H. Eles são capazes de se ligar a uma cadeia em formação ( possuem a extremidade 5’- trifosfato normal). Entretanto, uma vez incorporados a uma fita de DNA crescente o ddNTP não pode formar uma ligação fosfodiéster com o próximo dNTP a ser incorporado. Assim, a incorporação dos ddNTP na fita gera fragmentos de diferentes tamanhos (dependendo de qual posição o ddNTP foi incorporado) diferindo entre si por uma base e permite, após eletroforese, diagnosticar qual é a posição de cada base na fita de DNA.
Obtém-se uma amostra do fragmento de DNA a ser seqüenciado e em seguida amplifica-se este DNA utilizando dNTP’s normais e ddNTP’s marcados com fluorescência (sendo cada um dos 4 ddNTP’s diferentes apresenta fluorescência de uma cor). São gerados, então, conjuntos de moléculas que diferem em tamanho por uma base (o ddNTP marcado com fluorescência que se inseriu na ponta 3’). Realiza-se a eletroforese. A máquina de seqüenciamento automático detecta a fluorescência por leitura a laser, sendo que a primeira base/fragmento diagnosticado é complementar à primeira base da fita a ser seqüenciada e assim por diante.
Questão 4: Eva Katharina Barbosa Pfannes
Sabe-se que a taxa de mutação espontânea na replicação do DNA é de aproximadamente 10-7 . Mas, em E.coli , esta taxa é de 10-10 devido ao monitoramento pelo sistema de reparo, principalmente através da correção dos despareamentos após a replicação. Por que, se não houvesse a marcação do DNA velho com grupos metila , não seria possível o reparo por despareamento?
O que você esperaria que acontecesse se o DNA, em um teste in vitro, fosse exposto a enzimas do sistema de reparo por despareamento:
Resposta:
Na E. Coli, a metilação na posição N6 de todas as adeninas que ocorrem nas seqüências (5’) GATC permite a diferenciação entre a fita molde e a fita recém- sintetizada, durante um curto período no qual a fita nova ainda não foi metilada. Uma proteína do complexo de despareamento reconhece a fita recém-sintetizada não-metilada e outra proteína cliva o DNA para que o segmento despareado do DNA seja refeito pela DNA polimerase.
Se ambas as fitas fossem metiladas em uma seqüência GATC, os despareamentos não seriam reparados porque o complexo de reparo não reconheceria a fita recém-sintetizada. Se nenhuma fita estivesse metilada haveria o reparo, mas este não seria específico para nenhuma fita de DNA, podendo não haver correspondência entre a informação de uma fita ou de outra, já que não se identificaria uma fita molde para comparação da seqüência.
Questão 5:Paloma de Alvarenga Côrtes
Imagine que você precisa detectar uma proteína de vírus no sangue de camundongos. Cite e descreva a técnica que você utilizaria.
Resposta:
Transferência Western.
Nesta técnica, submete-se a amostra à eletroforese em um gel de SDS poliacrilamida .Após separadas no gel as proteínas são transferidas por contato ou por um campo elétrico para a superfície de uma lâmina de polímero. Adiciona-se um anticorpo específico para a proteína de interesse. Adiciona-se um segundo anticorpo marcado radiativamente
específico para o primeiro. Um auto-radiograma revela uma faixa escura onde está a proteína de interesse. Como alternativa, o segundo anticorpo pode conter uma enzima que gera um produto corado.
Questão 3: Camila Mendes de Sá
O DNA plasmidial de E. coli possui uma única origem de replicação, o locus oriC , que contém uma disposição atrelada de três blocos de 13 de seqüências ricos em pares de bases A-T, o que facilita a separação do dúplex para começar a síntese do DNA. Enquanto isso, o DNA eucariótico é replicado a partir de muitas origens, necessárias para que a replicação ocorra de forma rápida, devido ao grande comprimento dos DNAs eucarióticos. A ativação de cada ponto de início gera duas forquilhas divergentes de replicação, enquanto que em E. coli, uma forquilha é gerada.
Em E. coli, na forquilha de replicação, ambos os filamentos do DNA parental servem como moldes para a síntese do novo DNA. Uma significativa proporção de DNA recém—sintetizado existe como pequenos fragmentos (fragmentos de Okazaki), que estão presentes por curto tempo na vizinhança da forquilha. À medida que a replicação continua, estes fragmentos tornam-se covalentemente unidos pela DNA ligase para formar o filamento lagging; o outro filamento novo, o leading, é sintetizado continuamente. Os fragmentos de Okazaki e os de leading são sintetizados no sentido 5’ ® 3’. Uma reunião de multissubunidades contendo polimerase III sintetiza a maior parte do novo DNA, enquanto a a polimerase I apaga o primer e preenche os espaços. A DNA polimerase II participa do reparo do DNA, mas não é necessária para a replicação do DNA.
As polimerases a e d têm papel importante na replicação cromossômica de células eucarióticas e b e e participam no reparo do DNA. Estas enzimas, como as DNA polimerases de procariontes, usam desoxirribonucleosídeos trifosfatos como intermediários ativados e fazem o alongamento no sentido de 5’ para 3’. A síntese descontínua do filamento leading é catalisada por a , e a síntese contínua do filamento leading por d . O complexo a tem uma subunidade de primase que faz trechos temporários de RNA que sevem como primer e que são depois removidos. As enzimas d e b possuem atividade de exonuclease 3’ ® 5’ com capacidades de edição similares às das polimerases procarióticas. Uma proteína acessória provavelmente faz a revisão do DNA sintetizado por a .
GRUPO 3
Guimária Geraldina Goulart 2002024728
Como construir uma biblioteca genônica humana, usando como vetor o fago l?
Uma amostra do DNA genômico total é primeiro separada mecânicamente ou parcialmente digerida por enzimas de restrição em grandes fragmentos. Essa população quase aleatória de fragmentos de DNA superpostos é, então, separada por eletroforese em gel para isolar trechos com de 15 Kb de comprimento. Ligantes sintéticos são unidos às pontas desses fragmentos, são formadas pontas adesivas, eos fragmentos são, então, inseridos em um vetor, tal como o DNA do fagol, preparado com as mesmas pontas adesivas. As E.coli são, então, infectadas com esses fagos recombinates. O lisado resultante contém fragmentos de DNA humano abrigados em um número suficientemente grande de vírus para garantir que quase todo o genoma esteja representado. Estes fagos constituem uma biblioteca genômica.
Rafael Antonius Pfeilsticker de Knegt 2002025112
O que é necessário para a síntese de DNA e RNA? Qual a fonte de energia utilizada?
Para a síntese de DNA é necessário um DNA molde, um primer, dNTPs, enzimas( DNA polimerase dependente de DNA), sendo que a energia provém dos precursores (dNTPs), da clivagem entre o P beta e o gama. Já para RNA os precursores são o ATP, CTP, GTP,UTP, DNA molde, e RNA polimerase dependente de DNA, não é nescessário primer, sendo que a energia vem da liberação do pirofosfato e ligação do P alfa com o O da 3’.
Ivan Ávila Mortimer
Qual a temperatura utilizada na PCR? A que temperatura ela sintetiza DNA? Porque sintetiza DNA a tão alta temperatura?
É a enzima Taq polimerase. Ela sintetiza DNA a 72ºC. Ela sintetiza DNA a tão alta teperatura devido ao fato dela ser uma enzima de uma bactéria termófila aquática que vive em altas temperaturas.
Sibelle Torres Vilaça 2002025139
a)Explique qual é a vantagem de se ter timina ao invés de uracila no DNA.
b)Explique o mecanismo de reparo que retira as uracilas do DNA.
a) Caso houvesse uma uracila no DNA, com a desaminação espontânea de uma citosina, que se transforma em uracila, não seria possível identificar qual seria a fita a ser corrigida. Com a timina, a presença da uracila no DNA indica qual é a fita que deve ser reparada. Esse mecanismo de reparo de bases é mais importante no DNA, pois, ao contrário do RNA, esta é a molécula que é transmitida hereditariamente, enquando que o RNA suporta mais erros por não ser transmitida as demais gerações.
b)O reparo é feito através da excisão de base. As DNAs glicosilases reconhecem a C desanimada e clivam a ligação N-glicosídica (base-açúcar), liberando a base e gerando um sítio AP. Uma AP endonuclease corta a ligação fostodiéster. Uma exonuclease de excisão remove um trecho de DNA, a DNA polimerase sintetiza um novo trecho e por fim, a DNA ligase sela o corte
GRUPO 4
Determinados tipos de lesão no DNA impedem a transcrição travando a RNA polimerase. A enzima travada serve como sinal para o sistema de reparo.
As nucleases do DNA são enzima que degradam o DNA. Elas são divididas em duas grandes classes: as exonucleases, que degradam o DNA a partir das extremidades da molécula. Muitas operam apenas na direção 5`à 3` ou na direção 3`à 5`, removendo nucleotídeos apenas da extremidade 5`ou 3`de uma fita de ácido desoxirribonucléico. As endonucleases reconhecem seqüências específicas de bases na dupla hélice do DNA e clivam ambos os filamentos do dupléx em pontos específicos .
*Clonagem de DNA dependente de célula:
1º etapa- construção de DNA recombinante, sendo necessário o DNA alvo, o replicon (vetor), juntamente com as enzimas de restrição e DNA ligase.
2º etapa- as moléculas do DNA recombinante são transferidas para a célula hospedeira.
3º etapa- propagação seletiva de clones celulares
4º etapa- isolamento de clones
*Clonagem de DNA independente de célula: PCR
1º etapa- ocorre a separação dos filamentos do DNA
2º etapa- hibridação dos primers
3º etapa- síntese de DNA
Os reagentes da reação de PCR são: solução com a seqüência alvo, primers (forward e reverse), DNA polimerase (Taq polimerase), dNTPs.
O mismatch repair reconhece a fita nova devido esta não ser metilada. Ocorre uma excisão na fita nova e uma exonuclease degrada parte desta fita, logo em seguida uma DNA polimerase reconstitui a parte da fita que foi degradada fazendo o pareamento correto.
A reação de síntese da cadeia de DNA ocorre no sentido 5´à
3`, isto é, uma hidroxila ligada ao carbono 3` do desoxirribonucleotídeo da cadeia que esta sendo formada reage com o grupamento fosfato do desoxirribonucleotídeo que vai ser incorporado na cadeia. O grupo fosfato é composto de três átomos de P, ![]() ,
, ![]() e
e ![]() . Os átomos
. Os átomos ![]() e
e ![]() são perdidos na reação que produz a ligação fosfodiester, somente o átomo
são perdidos na reação que produz a ligação fosfodiester, somente o átomo ![]() fica ligado na cadeia de DNA, logo ele deve ser o átomo de fósforo a ser marcado radioativamente, conseqüentemente marcando a cadeia de DNA.
fica ligado na cadeia de DNA, logo ele deve ser o átomo de fósforo a ser marcado radioativamente, conseqüentemente marcando a cadeia de DNA.
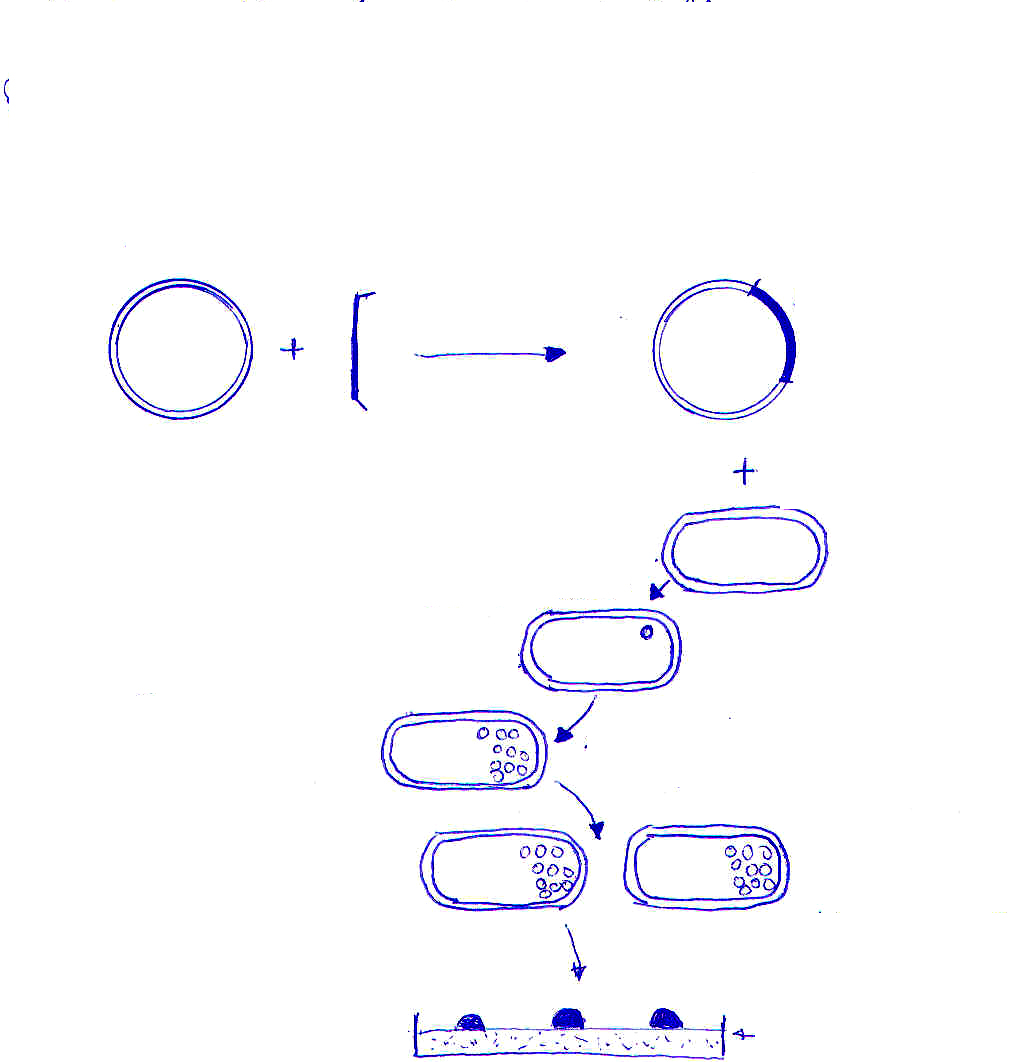
7. (Aluno: Saulo Augusto de P. Pinto) Sobre a replicação do DNA em eucariotos (mais especificamente em levedura), responda:
a) O que são e para que servem os fatores de licenciamento?
b) O que é o chaveamento de polimerases (polimerase switching). Quais as vantagens proporcionadas por esse processo?
RESPOSTAS:
a) Os fatores de licenciamento são proteínas recrutadas pelo complexo de origem de replicação (ORC). Elas permitem a formação do complexo de iniciação. Servem para garantir que cada replicon é replicado apenas uma vez durante o ciclo celular.
b) O chaveamento de polimerases é o nome dado à substituição da polimerase alfa pela polimerase delta durante a replicação. As vantagens são a alta processividade da polimerase delta em relação à alfa (longas seqüências podem ser replicadas sem interrupção), e a capacidade de executar a edição do DNA pois a polimerase delta possui atividade de exonuclease 3´à 5´.
GRUPO 5
Ralph Thomé
1) A maior estabilidade do DNA provavelmente é responsável por seu uso como material hereditário nas células modernas e em muitos vírus. A que se deve essa estabilidade?
Resposta: A ausência de grupo OH no carbono 2' da ose aumenta a resistência do DNA contra hidrólises.
Thiago Carvalho
2) O que marca o início de uma unidade transcricional?
Resposta: A fita de DNA molde contém regiões chamadas de promotoras onde especificamente se liga a RNA polimerase e determinam onde a transcrição começa.
Matheus Rajão
3) Qual a vantagem de se ter timina ao invés de uracila no DNA?
Resposta: A citosina é facilmente desaminada gerand uma molécula de uracila. Ao reparar esse erro, difere-se a fita velha da fita nova a partir da metila presente na timina ( e ausente na uracila). Se no DNA houvesse uracila, não haveria como descobrir qual base foi desaminada.
Jõao Pedro Gomes
4) Qual a razão de não se verificar uma maquinaria de reparo de RNA?
Resposta: O RNA não é utilizado para passar informações genéticas para uma próxima geração. E mesmo se houvesse um sistema de reparo (ou mesmo uma RNA polimerase com atividade "proffreading") no RNA, a transcrição - tradução de proteínas tornar-se-ia um processo lento e custoso.
Maurício Martins
5) Atualmente, a produção mundial de insulina para diabéticos e feita quase que exclusivamente por bactérias. Explique como isso é possível.
Resposta: Após a seqüência de DNA que codifica a insulina ter sido elucidada, essa foi clonada e inserida em um vetor (plasmídeo), criando dessa forma um fragmento de DNA recombinante. Esse plasmídeo é inserido em bactérias a fim de se produzir o hormônio em questão.
Flávia Pezzini
6) Quais são os elementos condicionantes para o trabalho da DNA polimerase I de sintetizar uma cadeia de DNA? De onde vem a energia para a polimerização? O que significa dizer que ela é uma enzima dirigira por molde?
A DNA polimerase I é uma cadeia polipeptídica única que catalisa a adição gradual de unidades desoxirribonucleotídicas à cadeia de DNA. Ela requer os seguintes componentes para sintetizar uma cadeia de DNA:
A reação de alongamento da cadeia catalisada pela DNA polimerase I é o ataque nucleofílico do grupamento terminal 3´- OH do primer ao átomo de fósforo mais interno de um desoxirribonucleosídeo trifosfato. Forma-se uma ponte fosfodiester, e o pirofosfato é concomitantemente liberado. A hidrólise subseqüente do pirofosfato pela pirofosfatase inorgânica fornece energia para a polimerização. O alongamento se faz no sentido de 5´para 3´.
A DNA polimerase I só catalisa a formação de uma ligação fosfodiester se a base do nucleotídeo que chega for complementar à base do filamento molde. Ou seja, ela é dirigida por molde. A enzima obtém instruções do molde e sintetiza um produto com uma seqüência de bases complementar à do molde. A DNA polimerase I pode também corrigir erros no DNA removendo nucleotídeos mal pareados. Assim, há apurada alta fidelidade da replicação do DNA.
GRUPO 6
Questão 1 - Igor Brant
Questão 2 - Josimar Gomes
Questão 3 - Flavio Henrique
1.1_ defina íntrons e éxons .
Íntrons : São os trechos de RNA que são retirados durante o processo de splicing, quando são recompostos os fragmentos que codificam os domínios de proteínas .
Éxons : São os trechos mantidos no mRNA e que na maioria das vezes codificam domínios de proteínas.
1.2_ Quais as vantagens de se ter genes contínuos ? e descontínuos?
Ter genes contínuos reduz muito o tamanho do material genético do organismo facilitando assim o crescimento e multiplicação rápida de organismos mais simples.
Já os genes descontínuos oferecem a possibilidade de rearranjo de éxons , que seria uma forma rápida e eficiente de criar novos genes . Uma outra vantagem é a potencialidade de se gerar uma série de proteínas relacionadas, pelo splicing feito de modos diferentes em um mRNA nascente. Com esse recurso um organismo complexo pode gerar várias proteínas diferentes a partir de um tema básico sem a necessidade de um gene para cada proteína .
1.3_Esses íntrons surgiram durante a evolução dos organismos complexos ou foram posteriormente perdidos pelos organismos simples ?
comparações das sequências de dna que codificam proteínas conservadas na evolução sugerem que os íntrons já estavam presentes nos genes ancestrais. Além disso um mecanismo comum de splicing deve Ter surgido ainda antes da divergência evolutiva entre fungos plantas e animais . Isso pôde ser observado em um experimento no qual extrato de células de mamíferos puderam recompor RNA de leveduras.
GRUPO 7
Questionário de Biologia Molecular
Questão 1. (Maíra Neves Santos)
Sobre a Taq polimerase, responda:
a-) Por que esta enzima é utilizada na PCR, em lugar da DNApolimerase de E.coli ?
Porque a DNApolimerase de E.coli é sensível ao calor, sendo destruída em temperaturas necessárias à separação das cadeias duplas de Dna. Já a Taq polimerase tem temperatura ótima de ação a 72° C e é razoavelmente estável a 94°C.
b-) Qual a taxa de incorporações de nucleotídeos errados por esta enzima? Qual o motivo desta taxa?
A Taq polimerase incorpora um nucleotídeo incorreto para cerca de 2 x 104 nucleotídeos incorporados (o que é alto em comparação com a taxa de erro na replicação natural da molécula de DNA, que é de 1 para 109). Isto ocorre porque a Taqpolimerase, in vitro, não tem capacidade de revisar a seqüência sintetizada e também é influenciada pelas temperaturas e concentração de sal, típicas da PCR.
Questão 2. (Rodrigo)
a-) Escreva como podem ser formadas as espécies ativas de oxigênio na célula.
A mitocôndria reduz o oxigênio à água, com a entrega de quatro elétrons em um único passo, no entanto, ela erra em 5% das vezes gerando as espécies ativas de oxigênio, por exemplo, superóxidos e H2O2 .
b-) Qual a interferência das espécies ativas de oxigênio na célula?
A H2O2 combina com FeII gerando radical hidroxila, lesionando o DNA que é quebrado em Fiat simples.
Questão 3. (Clarissa)
Qual a diferença entre os mecanismos de reparo NER e BER?
O NER (nucleotide excision repair) reconhece a distorção do DNA e retira alguns nucleotídeos. Já BER (base excision repair) a glicosilase reconhece a base trocada e retira quebrando a ligação glicosídica entre a base e a ribose. Gera-se um sítio abásico, que é substrato para a AP-endonuclease. A DNA polimeraseI e a DNA ligase finalizam o reparo.
Questão 4. (Guilherme Trópia)
A manutenção da estabilidade genética necessita não apenas de um mecanismo extremamente preciso para replicação do DNA, antes da célula se dividir, mas também de mecanismos para reparação das muitas lesões acidentais que ocorrem continuamente no DNA.
Responda:
a-) Cite três causas dessas lesões que ocorrem no DNA.
O DNA sofre grandes mudanças como resultados de flutuações térmicas, cerca de 5000 bases purinas são perdidas por dia no DNA de cada célula humana devido a disrupção térmica de suas ligações N-glicosílicas à desoxirrbose. A desaminação espontânea de citosina a uracil, no DNA, é estimada a uma taxa de 100 bases por genoma/dia. As bases do DNA também estão sujeitas a mudanças por metabólitos reativos, que podem alterar a capacidade de pareamento, e pela luz ultravioleta do sol, que promove a ligação covalente entre duas pirimidinas adjacentes no DNA.
b-) Descreva a via básica para a reparação do DNA.
A porção alterada da fita de DNA danificada é reconhecida e retirada por enzimas denominadas nucleases de reparação do DNA, que hidrolizam as ligações fosfodiéster que mantêm os nucleotídeos danificados ligados ao resto da molécula de DNA, deixando nessa região, uma pequena falha na hélice de DNA. Uma outra enzima, a DNA polimerase, liga-se à extremidade 3’-OH da fita de DNA cortada e preenche a falha fazendo uma cópia complementar da informação contida na fita intacta. A quebra na fita danificada, deixada quando a DNA polimerase preencheu a falha, é restabelecida por um terceiro tipo de enzima, a DNA ligase, que completa o processo de restauração.
Questão 5. (Nara Mota)
Um plasmídio de clonagem deve ter pelo menos certas características que facilitem a clonagem e que permita avaliar a qualidade da biblioteca genômica com ele construída. Quais são essas características?
Um sítio de restrição único para determinada enzima, localizado na ORF de um gene de resistência a uma droga A, um segundo gene de resistência par uma droga B e uma origem de replicação.
Questão 6. (Ludmila)
O que aconteceria se a menor temperatura de um ciclo de PCR for reduzida de 56oC para 45oC? Explique.
Outras bandas apareceram no gel como resultado de hibridização dos primers com novos sítios, com baixa estringência.