Saldo de Bases
Rodando Phred com a chance de erro indicada (eixo do X), o saldo de bases consideradas de boa leitura em cada ponto (eixo do Y) foi avaliado para cada uma das sequências. O resultado do BLAST contra a sequência publicada do vetor foi utilizado como parâmetro de referência. Compare a perda de bases observada com erro de 1% (Phred 20, média acusando perda de 273 bases) ao observado com erro de 10% (Phred 10, média acusando perda de 92 bases). Com chance de erro de 16%, a média acusa nem perda nem ganho de bases, mas a amplitude da distribuição é grande, havendo grande incorporação de bases incorretamente nomeadas a muitas leituras. Não há aumento significativo de incorporação de bases erradas até a chance de erro de 14%, onde há uma perda média de 43 bases.
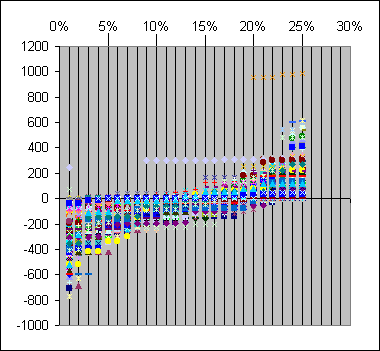 Legenda: Plasmídio com seqüência conhecida foi submetido a seqüenciamento em três tubos e o material reunido e distribuído em placas de 96. O arquivo processado .esd produzido pelo MegaBACE foi utilizado para rodar o programa Phred utilizando o algoritmo -trim_alt calibrado para trimar a seqüência com a freqüência de erro indicada no eixo dos X (1 a 25%). As seqüencias não trimadas foram submetidas a pesquisa de homologia com BLASTn contra a seqüência conhecida do vetor. As posições iniciais e finais dos alinhamentos foram usadas como referência. O saldo de bases, nas duas extremidades, foi calculado com referência ao resultado do BLAST. Saldo negativo indica que a trimagem foi exagerada, não aproveitando bases presentes no alinhamento, enquanto saldo positivo devem representar bases espuriamente nomeadas e não trimadas.
Legenda: Plasmídio com seqüência conhecida foi submetido a seqüenciamento em três tubos e o material reunido e distribuído em placas de 96. O arquivo processado .esd produzido pelo MegaBACE foi utilizado para rodar o programa Phred utilizando o algoritmo -trim_alt calibrado para trimar a seqüência com a freqüência de erro indicada no eixo dos X (1 a 25%). As seqüencias não trimadas foram submetidas a pesquisa de homologia com BLASTn contra a seqüência conhecida do vetor. As posições iniciais e finais dos alinhamentos foram usadas como referência. O saldo de bases, nas duas extremidades, foi calculado com referência ao resultado do BLAST. Saldo negativo indica que a trimagem foi exagerada, não aproveitando bases presentes no alinhamento, enquanto saldo positivo devem representar bases espuriamente nomeadas e não trimadas.

