kit PCR-Select cDNA subtraction
Duas populações de mRNA podem ser comparadas por técnicas subtrativas, que realçam as diferenças entre elas. Assim, são clonados genes que são expressos em uma situação e não em outra. Embora existam vários métodos, a teoria por trás deles é simples. Primeiro, ambas populações são convertidas a cDNA. Nesta técnica que vamos descrever, é comum chamarmos de "tester" a população de cDNA que apresenta alguns genes especificamente expressos e o cDNA de referência é chamado de "driver". Tester e driver são hibridizados e as seqüências híbridas são removidas. Consequentemente, o cDNA que não hibridizou representa genes que são expressos no tester e não no driver.
BASE MOLECULAR DA SUBTRAÇÃO
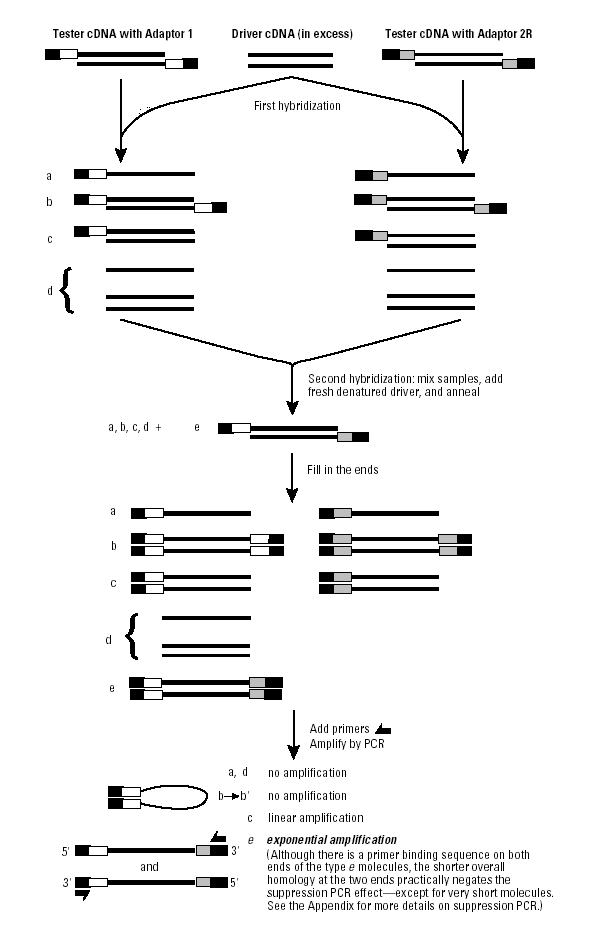
O procedimento está mostrado na figura. Primeiro o extrai-se RNA total das duas amostras e purifica-se com resina de oligo-dT o RNA poly A+, enriquecido portando de mRNA. O cDNA é sintetizado a partir do mRNA (é necessário 0,5 - 2
mg). Digere-se o cDNA tester e driver com uma enzima de restrição que corta com alta frequência, pois seu sítio de restrição exige apenas o reconhecimento de 4 bases (enzima Rsa I por exemplo) e que produz extremidades cegas (as duas fitas têm o mesmo comprimento). Divide-se agora o cDNA tester em duas porções e cada uma é ligada a um adaptador (uma pequena molécula de DNA dupla fita) diferente. Como os adaptadores utilizados não contêm fosfato presente na extremidade 5', apenas uma de suas fitas é covelentemente ligada ao cDNA. Em outras palavras, permite-se somente que a fita "watson" se ligue às extremidades 5' do cDNA (portanto uma fita de adaptador em cada extremidade do cDNA). As duas preparações de tester são hibridizadas, separadamente, com excesso de driver, de forma que o cDNA que existe somente nas preparações tester irá permanecer simples fita. Uma segunda hibridização é feita misturando-se as duas preparações sem desnaturá-las, para que as fitas exclusivas do tester de uma preparação hibridizem com as fitas exclusivas do tester da outra. Para garantir, adiciona-se ainda uma quantidade de driver frescamente desnaturado para impedir a dimerização de moléculas dos dois tipos de driver que não são diferencialmente expressas. Incuba-se a mistura agora com DNA polimerase para que haja síntese das fitas "crick" dos adaptadores. Serão produzidas moléculas com adaptadores nas duas pontas (adaptadores iguais ou diferentes). Agora um detalhe sobre os dois diferentes adaptadores: a metade 5' (a cauda) de ambos é idêntica, mas a outra metade difere. Isso é importante, pois quando o mesmo adaptador está nas duas extremidades, forma-se uma alça pela hibridização do adaptador das duas extremidades; todavia, quando são adaptadores diferentes, somente a metade deles é idêntica e na temperatura de incubação não é possível mantê-los ligados, não havendo formação da alça. Utilizando agora iniciadores que com a seqüência que é idêntica em ambos tipos de adaptador, é possível a amplificação por PCR das moléculas que contém um tipo de adaptador em cada extremidade. Note que essa amplificação foi gerada por moléculas presentes nas duas preparações de tester que não hibridizaram com o cDNA driver e puderam portanto hibridizar entre si. Portanto, representam moléculas ausentes no driver. Em seguida à primeira PCR, é realizada outra com iniciadores mais internos, que precisam hibridizar simultaneamente em ambas extremidades, iniciadores equivalentes portanto às regiões diferentes dos dois adaptadores.Para clonar os produtos de PCR, pode-se digerir os mesmos com as enzimas de restrição cujos sítios estão presentes nos dois adaptadores: Not I em um adaptador e Eag I no outro adaptador. Ou então pode-se digerir o cDNA com Rsa I, sítio que foi regenerado quando o adaptador foi ligado ao cDNA. Após clonados, os cDNA podem ser seqüenciados e seus produtos identificados por pesquisa de homologia com seqüências presentes em bases de dados.
CONFIRMAÇÃO DA EXPRESSÃO DIFERENCIAL
Geralmente é feita por Northern Blot em etapa final, mas uma análise prévia pode ser feita utilizando-se slot blot onde os vários clones são depositados sobre um filtro e cDNA das duas situações são marcados radioativamente e utilizados na hibridização.
Uma outra variação é interessante, invertendo-se as posições de tester e driver, pois podem haver genes reprimidos pelo tratamento estudato.
