AULA RNAseq
Primeira parte: MONTAGEM E ANÁLISE DE EXPRESSÃO
1° Passo: atenção - login hj será bioinfo2 (agostinhocarrara) e não bioinfo (taxiscarrara)
Ao entrar na bioinfo, é possível ver entre outras pastas:
- anaconda3: pasta contendo todo o ambiente dos programas que serão utilizados nesta aula;
- data: pasta com todos os arquivos de entradas necessários para realizarmos a montagem dos transcritos.
Vamos criar então o nosso local de trabalho com o comando mkdir:
mkdir <seunome>
2° Passo:
Vamos copiar todos os arquivos necessários para podermos realizar esta prática. Para isso, vamos para o seu local de trabalho:
cd <seunome>
Agora vamos copiar os arquivos da pasta data, a um diretório acima do seu:
cp -r ../data/* .
Muito importante o ponto final depois do espaço depois do asterisco! Dá um ls
Dentro de sua pasta é possível encontrar os seguintes arquivos:
- rnaseq_data: pasta - com todas as reads que serão utilizadas no Trinity!
- uniprot_sprot.pep: arquivo - contendo os fastas de proteínas do banco de dados Uniprot;
- uniprot_sprot.dmnd: arquivo database já formatada para o Diamond usar, a partir dos fastas do Uniprot acima;
- diamond: alinhador tipo blastp que iremos utilizar para anotar os transcritos montados;
- samples.txt: arquivo que descreve as amostras e as condições utilizadas pelo experimento necessário para as comparações de expressão;
- filtrar.sh: um scriptzinho para poder pegar as sequencias dos genes, lá no final.
3° Passo:
Nesta aula utilizaremos dados de RNA-Seq correspondentes ao fungo Schizosaccharomyces pombe, contendo reads de 76 pares de base, paired-end, correspondentes a duas amostras: Sp_log (crescimento logarítmico) e Sp_plat (fase de platô). Dentro da pasta RNASEQ_data temos essas reads, e é possível encontrar os arquivos com final left.fq e right.fq no formato FASTQ, saída ao estilo do sequenciador Illumina:
Para poder realizar a montagem dos transcritos (leva 16 min), vamos utilizar todas as reads para montar um único arquivo fasta contendo todos os transcritos montados. Isto é feito para futuramente utilizarmos este arquivo para realizar a análise de genes diferenciais. Iremos rodar o Trinity com o seguinte comando (sopie e cole, depois explicamos):
---------------------------------------------------------------------------------------------------------------------------
Trinity --seqType fq --SS_lib_type RF --left rnaseq_data/Sp_log.left.fq.gz,rnaseq_data/Sp_plat.left.fq.gz
--right rnaseq_data/Sp_log.right.fq.gz,rnaseq_data/Sp_plat.right.fq.gz
--CPU 1 --max_memory 1G --output trinity_saida/
---------------------------------------------------------------------------------------------------------------------------
Detalhes:
- --left (rnaseq_data/Sp_log.left.fq.gz,rnaseq_data/Sp_plat.left.fq.gz): indicamos o caminho de todas as reads no sentido forward, tanto log quanto plat;
- --right (rnaseq_data/Sp_log.right.fq.gz,rnaseq_data/Sp_plat.right.fq.gz): indicamos o caminho de todas as reads no sentido reverse;
- Não se esqueça de que estes arquivos devem ser separados por vírgulas, e não espaçamentos.
- --SS_lib_type (RF): seleciona o tipo da sua biblioteca, se é paired-end (reverse+forward) ou single-end;
- --CPU (1): quantidade de threads que iremos utilizar;
- --max_memory (1G): quantidade máxima de memória que o Trinity irá utilizar;
- --output (trinity_saida/): local no qual o Trinity irá salvar todos os dados relacionados à montagem dos transcritos. A barra no fim diz que vai ser um diretório!
- Importante que esta pasta esteja criada nomeada como trinity_saida para o resto funcionar.
Após completar a montagem, será criada a pasta chamada trinity_saida, tal como o seu output foi descrito. Dentro desta pasta é possível encontrar todos os dados relacionados à montagem dos transcritos.
4° Passo:
Vamos agora verificar o arquivo de montagem dos transcritos com os comandos:
cd trinity_saida ou pwd pois acho que vc já está na pasta!
less Trinity.fasta
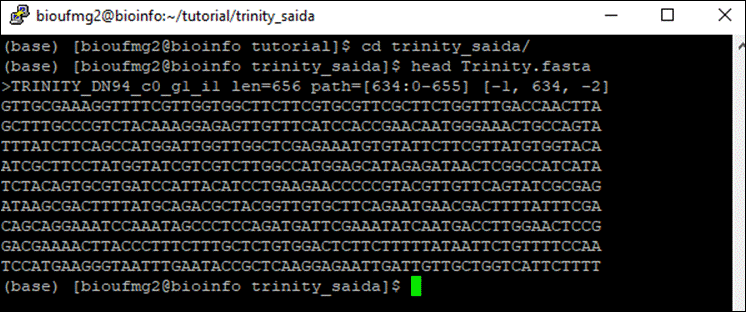
Informações importantes:
- O identificador > é utilizado em todo arquivo fasta, para ser um cabeçalho para a sequência;
- TRINITY_DN94, como no exemplo, é o nome do transcrito montado;
- _g1: indica que é o gene 1 montado;
- _i1: indica que é a isoforma 1 do gene 1 montado (ele pode ou não montar mais que uma isoforma do mRNA).
CURIOSIDADE:
Como os transcritos são processados em forma de gráfos de De Bruijn, nós podemos visualizar utilizando o programa Bandage (https://rrwick.github.io/Bandage/).
Vídeo explicativo caso queira tentar em casa em: https://www.youtube.com/watch?v=VuRN28XyFcI
5° Passo:
Vamos analisar agora os dados relacionados a esta montagem utilizando um script do Trinity chamado TrinityStats.pl. Para isso, vamos para a pasta do trinity_saida:
cd trinity_saida
Agora digite o comando para executar o script:
TrinityStats.pl Trinity.fasta
Resultado:
################################
## Counts
of transcripts, etc.
################################
Total
trinity 'genes': 396
Total trinity transcripts: 400 <<< logo 4 genes teriam 2 isoformas cada
Percent GC:
39.16
########################################
Stats based
on ALL transcript contigs:
########################################
Contig N10: 2589
Contig N20: 2148
Contig N30: 1818
Contig N40: 1594
Contig N50:
1329
Median contig
length: 548.5
Average contig:
839.30
Total assembled bases: 335720
#####################################################
## Stats
based on ONLY LONGEST ISOFORM per 'GENE':
#####################################################
Contig N10: 2589
Contig N20: 2118
Contig N30: 1818
Contig N40: 1584
Contig N50: 1322
Median contig length: 545.5
Average contig: 830.32
Total assembled bases: 328805
Neste reporte é possível ter acesso a informações como: número de genes encontrados, número de transcritos, a porcentagem de GC global e a média do tamanho dos contigs.
- Nx: Todos os contigs são ordenados, a partir do seu tamanho, do maior para o menor; o menor contig montado dentre os maiores compreendendo x% dos nucleotídeos totais, representa o seu Nx:
Exemplo com transcrito 2 ocorrendo em 50% das bases totais ordenadas:
1111111111111111111111111222222222222222233333333333444444455555
6° Passo:
Vamos agora contar a abundância de cada transcrito e sumarizar para gene que tenha isoformas, que foram montados utilizando o Salmon em cada condição com os seguintes comandos mas:
Volte agora para o seu diretório de trabalho:
cd ..
E digite os comandos (olha bem, são dois!):
---------------------------------------------------------------------------------------------------------------------------
/home/bioufmg2/anaconda3/pkgs/trinityrnaseq-v2.12.0/util/align_and_estimate_abundance.pl
--seqType fq --left
rnaseq_data/Sp_log.left.fq.gz --right rnaseq_data/Sp_log.right.fq.gz --transcripts trinity_saida/Trinity.fasta --est_method salmon --trinity_mode --prep_reference --output_dir salmon_saida/sp_log/
---------------------------------------------------------------------------------------------------------------------------
/home/bioufmg2/anaconda3/pkgs/trinityrnaseq-v2.12.0/util/align_and_estimate_abundance.pl
--seqType fq --left rnaseq_data/Sp_plat.left.fq.gz
--right rnaseq_data/Sp_plat.right.fq.gz --transcripts trinity_saida/Trinity.fasta --est_method salmon --trinity_mode --prep_reference --output_dir salmon_saida/sp_plat/
---------------------------------------------------------------------------------------------------------------------------
Detalhes (foi rápido né?):
- align_and_estimate_abundance.pl: script do pacote Trinity que ira realizar todo o pipeline;
- --seqType (fq): indicamos qual o formato dos arquivos das reads;
- --left (rnaseq_data/Sp_log.left.fq.gz): indicamos o caminho de todas as reads no sentido forward;
- --right (rnaseq_data/Sp_log.right.fq.gz): indicamos o caminho de todas as reads no sentido reverse;
- --transcripts (trinity_saida/Trinity.fasta): o caminho do arquivo contendo todos os transcritos montados pelo Trinity;
- --est_method (salmon): indicamos qual o método/software de estimação iremos utilizar;
- --trinity_mode: irá gerar um arquivo que irá mapear todas as isoformas em seus respectivos genes;
- --prep_reference: irá criar índices no arquivo dos transcritos para agilizar os processos;
- --output_dir (salmon_saida/sp_log ou sp_plat): indicamos qual o local dos arquivos de saída.
Ao final do processo, irá ser criada uma pasta chamada salmon_saida com duas pastas: Sp_log e Sp_plat, contendo todos os resultados obtidos pelo Salmon:
7° Passo:
Vamos para a pasta contendo os resultados de uma das condições para podermos analisar os arquivos de saída gerados pelo Salmon:
cd salmon_saida/sp_log (ou cd salmon_saida seguido de cd sp_log)
Dentro da pasta podemos encontrar 2
arquivos importantes, o quant.sf
e o quant.sf.genes
- quant.sf: quantificação da abundância dos transcritos;
- quant.sf.genes: quantificação de abundância a nível de genes (sumariza aqueles 4).
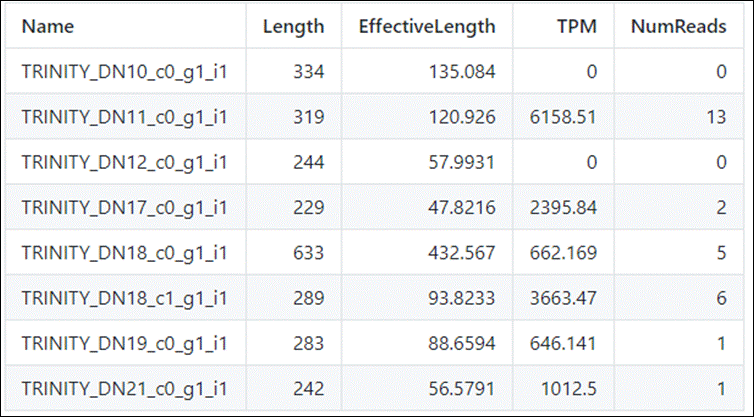
Estes arquivos são separados por tabulações e possuem colunas das quais podemos identificar o nome do transcrito/gene, o tamanho, e os valores de TPM e de suas reads (Transcritos Por Milhão, uma pormilhãonagem ao invés de porcentagem):
Utilizando o comando:
less quant.sf.genes
Obtemos o seguinte resultado (não se preocupe se o seu resultado estiver diferente da imagem, o importante são as colunas e seus valores):
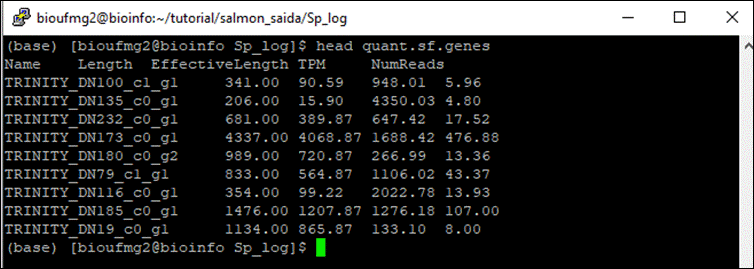
O gene DN173 está bem expresso ai e o DN135 tem o menor TPM.
8° Passo:
Agora vamos comparar os genes e transcritos entre as amostras log e plat utilizando um script perl do pacote Trinity chamado: abundance_estimates_to_matrix.pl. Primeiramente vamos para a nossa pasta de trabalho subindo dois diretórios (dê um pwd depois):
cd ../../ (ou cd
.. duas vezes, atenção, pwd tem que mostrar a pasta
com seu nome)
Este script irá calcular tanto a nível de genes tanto quanto de transcritos a abundância com o seguinte comando:
---------------------------------------------------------------------------------------------------------------------------
/home/bioufmg2/anaconda3/pkgs/trinityrnaseq-v2.12.0/util/abundance_estimates_to_matrix.pl --est_method
salmon --out_prefix salmon_saida/salmon
salmon_saida/sp_log/quant.sf salmon_saida/sp_plat/quant.sf --gene_trans_map trinity_saida/Trinity.fasta.gene_trans_map --name_sample_by_basedir
---------------------------------------------------------------------------------------------------------------------------
Detalhes:
- --est_method (salmon): indicamos qual o software de estimação foi utilizado para o cálculo de abundância;
- --out_prefix (salmon_saida/salmon): indicamos a parta e o nome dos arquivos de saída;
- --gene_trans_map (trinity_saida/Trinity.fasta.gene_trans_map): um índice contento cada transcrito associado com as suas isoformas gerado no passo 5;
- --name_sample_by_basedir: utilizado para poder nomear cada condição de acordo com o nome da pasta que as contém, ou seja, com a condição experimental.
Agora na pasta salmon_saida podemos ver que apareceram novos arquivos:
cd salmon_saida e ls
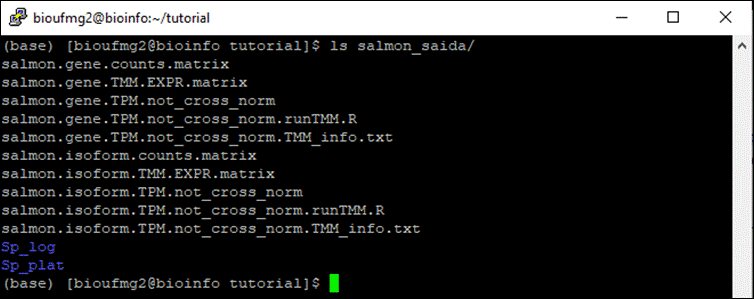
Arquivos importantes:
- Aqueles que possuem isoform são os resultados a nível dos transcritos e suas isoformas e aqueles que possuem gene são a nível de genes (agrupa isoformas);
- Arquivos terminados em .counts.matrix possuem a estimativa da contagem dos fragmentos de RNA-Seq;
- .TPM.not_cross_norm: matriz contendo os valores de expressão TPM que não estão normalizadas por amostra com genes muito expressos;
- .TMM.EXPR.matrix: matriz contendo os valores de expressão TMM normalizados.
Vamos para a salmon_saida:
cd salmon_saida (se vc ainda não estiver nela, olhe seu cursor ou dê pwd)
E digitar:
less
salmon.gene.counts.matrix
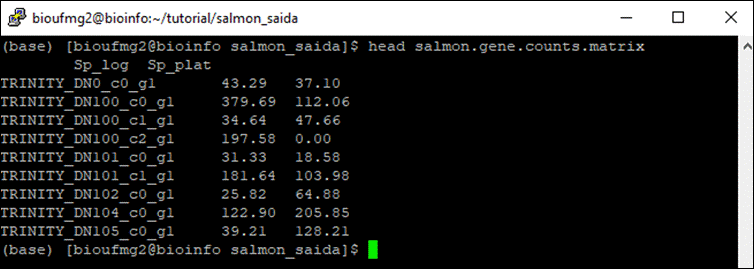
Podemos observar que as colunas dos valores foram nomeadas de acordo com a pasta em que elas estavam, facilitando o trabalho; nos próximos passos, para identificar qual dado é de qual amostra é importante. Chegaremos lá!
Também fique a vontade para ver os outros arquivos e perceber que todos eles seguem o mesmo padrão.
9° Passo:
Partiu agora descobrir os genes que estão diferencialmente expressos e montar uns gráficos para melhor visualização destes dados que criamos. Vamos utilizar o programa edgeR para este cálculo e outro script do Trinity chamado run_DE_analysis.pl.
Vamos agora sair da pasta do salmon_saida e vamos para a nossa pasta de trabalho:
cd ..
Certifique-se que está na sua pasta de trabalho para evitar problemas. Caso esteja perdido, utilize o comando pwd para ver o local do seu terminal.
Bora rodar:
---------------------------------------------------------------------------------------------------------------------------
/home/bioufmg2/anaconda3/pkgs/trinityrnaseq-v2.12.0/Analysis/DifferentialExpression/run_DE_analysis.pl
--matrix salmon_saida/salmon.gene.counts.matrix --method
edgeR --output edgeR_saida/ --samples_file samples.txt --dispersion 0.1
---------------------------------------------------------------------------------------------------------------------------
Detalhes:
- --matrix (salmon_saida/salmon.gene.counts.matrix): indicamos de qual matriz contém os dados de abundâncias dos transcritos de genes. Neste caso, iremos calcular somente os genes diferenciados e não suas isoformas.
- --method (edgeR): indicamos qual programa usar;
- --samples_file (samples.txt): um arquivo que descreve as replicatas e condições dos seus experimentos. Já já iremos vê-lo;
- --dispersion (0.1): parâmetro utilizado na função binomial negativa do edgeR para estimar a contagem dos genes.
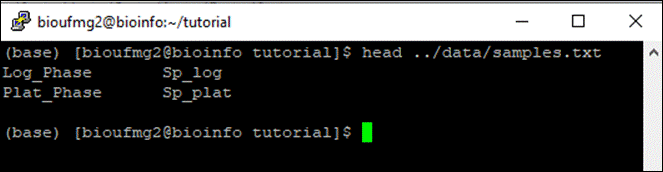
O arquivo samples.txt é muito importante nessa etapa uma vez que ele indica qual a condição e suas replicatas para o programa. Ele possui somente duas colunas, com suas linhas separadas por tabulações, e os nomes destas colunas devem estar de acordo com o nomes do passo 7, dentro do arquivo que possui o final .counts.matrix. Exemplo:
#condition #samples
condA
repA1
condA repA2
condB repB1
condB repB2
10° Passo:
Com os cálculos do edgeR realizados, vamos agora ver os arquivos de saída. Vamos para a pasta do edgeR_saida:
cd edgeR_saida
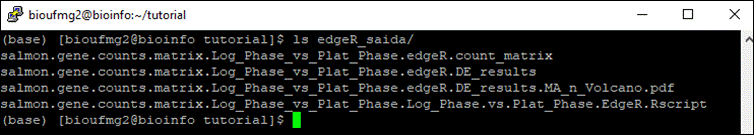
Arquivos importantes:
- O nome do arquivo segue uma lógica: {prefix}.sampleA_vs_sampleB.{method};
- .DE.results: arquivo contendo os resultados das análises, incluindo o fold change e a significância estatística (FDR);
- .MA_n_Volcano.pdf: nossos gráficos juntos, sendo um de MA e um Volcano!
Para ficar facilitar a visualização no navegador, digite um comando para simplificar o nome dele e escrever ele na sua pasta (um diretório acima) assim:
---------------------------------------------------------------------------------------------------------------------------
cp salmon.gene.counts.matrix.Log_Phase_vs_Plat_Phase.edgeR.DE_results.MA_n_Volcano.pdf
../MA_Volcano.pdf
---------------------------------------------------------------------------------------------------------------------------
Abra agora uma nova página no seu navegador no link:
http://bioinfo.icb.ufmg.br/bioufmg/look/
Será necessário um login (bioufmg) e senha (carrarataxis). Ao acessar, entre na pasta look e depois na pasta com o seu nome.
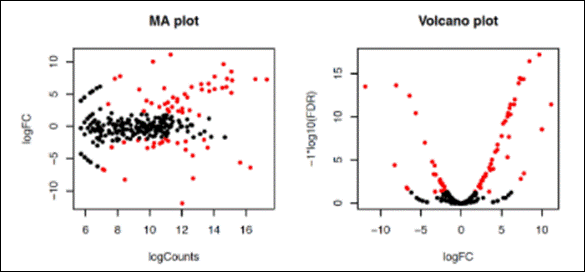
Ao abrir o arquivo PDF (MA_Volcano.pdf) podemos verificar que
as contagensas com um valor de FDR < 0.5
(os bãos de olhar) estão coloridas de
vermelho. FOld change expressa o número de dobradas ou caídas pela metade:
dobrar é 1, 2 é quadruplicar...
11° Passo:
Quantos genes estão diferencialmente expressos? Vamos descobrir com o seguinte comando (dentro da pasta edgeR_saida! pwd se estiver perdido):
---------------------------------------------------------------------------------------------------------------------------
cat
salmon.gene.counts.matrix.Log_Phase_vs_Plat_Phase.edgeR.DE_results | awk '{ if ($7 <= 0.05) print;}' | wc -l
---------------------------------------------------------------------------------------------------------------------------
Este comando irá quantificar quantos genes possuem um valor de FDR < 0.05 (coluna 7). Sinta-se livre para brincar com este valor.
12° Passo:
Vamos agora pegar todas as sequências de todos os genes que possuem o FDR <= 0.05 utilizando o script filtrar.sh. Para isso, vamos voltar para a nossa pasta de trabalho:
cd ../
Agora só rodar:
sh filtrar.sh
Após dar um ls, é possível ver que foram criados 2 arquivos:
DE_genes.txt: contém o nome de cada gene diferenialmente expresso;
DE_genes.fasta: contém o cabeçalho e a sequência do gene (o fasta);
2° PARTE: ANOTAÇÃO E EXPLORAÇÃO DOS GENES DIFERENCIALMENTE EXPRESSOS
COM BIOLOGIA DE SISTEMAS
1° Passo:
Iremos agora extrair esses genes e gerar os famosos gráficos de heatmap. Para isso utilizaremos o analyze_diff_expr.pl do Trinity, mas precisamos estar dentro da pasta edgeR_saida:
cd edgeR_saida/
---------------------------------------------------------------------------------------------------------------------------
/home/bioufmg2/anaconda3/opt/trinity-2.1.1/Analysis/DifferentialExpression/analyze_diff_expr.pl -P
1e-3 -C 2
--matrix ../salmon_saida/salmon.gene.TMM.EXPR.matrix
--samples ../samples.txt
---------------------------------------------------------------------------------------------------------------------------
Detalhes:
- -P (1e-3): define o p-valor a ser utilizado para ser considerado um gene diferencialmente expresso;
- -C (2): define o valor do fold change (quadruplica ou cai abaixo de ¼);
- --matrix (../salmon_saida/salmon.gene.TMM.EXPR.matrix): indicamos de qual matriz contém os dados de abundâncias dos transcritos de genes gerados nos passos anteriores contendo os valores de TMM entre as amostras;
- --samples (../samples.txt): aquele mesmo arquivo que descreve as replicatas e condições do seus experimentos.
Dando um ls nós vemos que arquivos novos que apareceram:
Arquivos importantes:
- .{sampleA}-UP.subset: features que estão reguladas positiviamentes na amostra A;
- .{sampleB}-UP.subset: features que estão reguladas positiviamentes na amostra B;
- diffExpr.{pvalor}_{valorFC}.matrix.log2.dat: todas a features encontradas que estão diferencialmente expressas em todas as comparações entre as amostras;
Vamos agora brincar com estes arquivos.
2° Passo:
Vamos
conhecer os genes super (UP) regulados em Plat_Phase sendo o p-valor menor
que 0,001 e mais que o dobro de valor. Repare na coluna 4 (Fold Change)
---------------------------------------------------------------------------------------------------------------------------
less salmon.gene.counts.matrix.Log_Phase_vs_Plat_Phase.edgeR.DE_results.P1e-3_C2.Plat_Phase-UP.subset
---------------------------------------------------------------------------------------------------------------------------
Fique a vontade para conhecer os
genes upregulated
da outra amostra!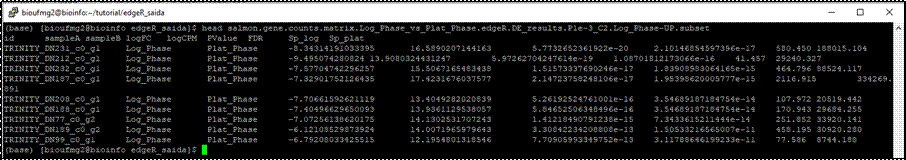
3° Passo:
Volte à sua pasta com cd .. e cd .. vamos analisar os fastas de DE_genes.fasta
Vamos
utilizar o Diamond
(tipo um blastx acelerado programaticamente) para
poder identificar cada gene para podermos conhecê-los. Para isso, certifique-se
que esteja na sua pasta de trabalho. Caso esteja perdido: pwd
Para rodar o Diamond, rode o seguinte comando:
---------------------------------------------------------------------------------------------------------------------------
Diamond blastx -d ../data/uniprot_sprot.dmnd -q DE_genes.fasta
-v --outfmt 6 -e 1e-10 --max-target-seqs
1 --outfmt 6 -o blastx.outfmt6
-b4
---------------------------------------------------------------------------------------------------------------------------
Detalhes:
- Estamos rodando o blastx do Diamond;
- -d (../data/uniprot_sprot.dmnd): indicamos o caminho do banco de dados que será utilizado para blastar as sequências. Neste caso, estamos utilizando o SwissProt-Uniprot;
- -q (DE_genes.fasta): indicamos a query, que neste caso é o arquivo que contém a sequência dos genes que possuem um valor de FDR <= 0.05;
- -v: modo verbose, para poder mostrar letrinhas no terminal;
- -e (1e-10): valor de e-value;
- --max-target-seqs (1): para poder anotar somente o best-hit de cada transcrito;
- --outfmt (6): define o modelo do qual será o arquivo de saída, que neste caso, é igual ao formato de saída do programa BLAST;
- -o (blastx.outfmt6): arquivo de output.
- -b4: argumento para poder não limitar a memória no uso do Diamond.
4° Passo:
Vamos agora conhecer os nossos transcritos a partir do arquivo de saída blastx.outfmt6. Se dermos um less neste arquivo podemos perceber que é um arquivo texto tabulado:
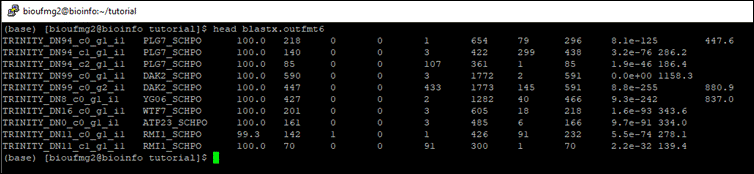
Nesse caso temos as seguintes colunas, como no BLAST:
- Coluna 1: Nome da query;
- Coluna 2: Nome da referência encontrada pelo transcrito (hit);
- Coluna 3: porcentagem de identidade;
- Coluna 4: tamanho do alinhamento;
- Coluna 5: número de mismatches;
- Coluna 6: número de gaps;
- Coluna 7; posição de início na sequência da query;
- Coluna 8: posição final na sequência da query;
- Coluna 9: posição de início na sequência da referência;
- Coluna 10: posição final na sequência da referência;
- Coluna 11: e-value;
- Coluna 12: bit score.
5° Passo:
Vamos agora identificar quais genes essas proteínas pertencem utilizando a plataforma online do Uniprot na aba de Get Gene Name. Primeiramente vamos printar somente a segunda coluna do nosso blast, referente aos nomes das proteínas:
awk '{print $2}'
blastx.outfmt6
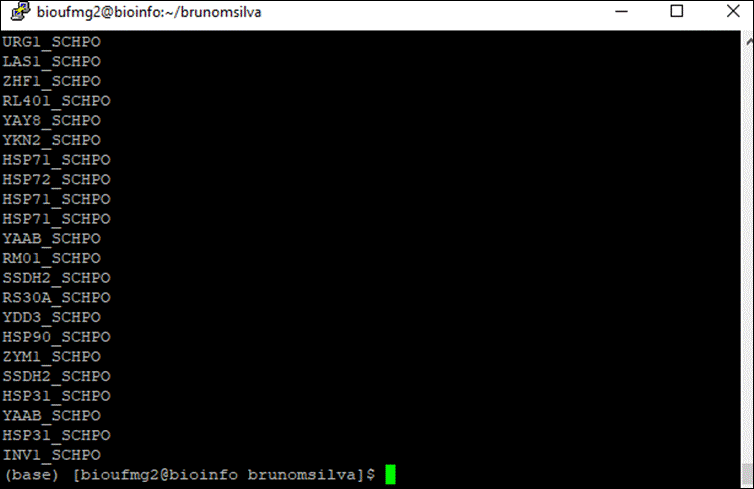
Selecione todas as linhas contendo estes nomes:
Lembrando que o PuTTy copia automaticamente ao selecionar alguma coisa no terminal.
6° Passo:
Acesse agora, numa nova aba do seu navegador, o site uniprot.org.
Logo no cabeçalho do site vamos clicar em ID mapping:
Cole o texto que você copiou do PuTTY (ctrl + v) na região do Enter one of more IDs; selecione, na parte de Select Options, To Gene Name. Por fim, clique em Submit.
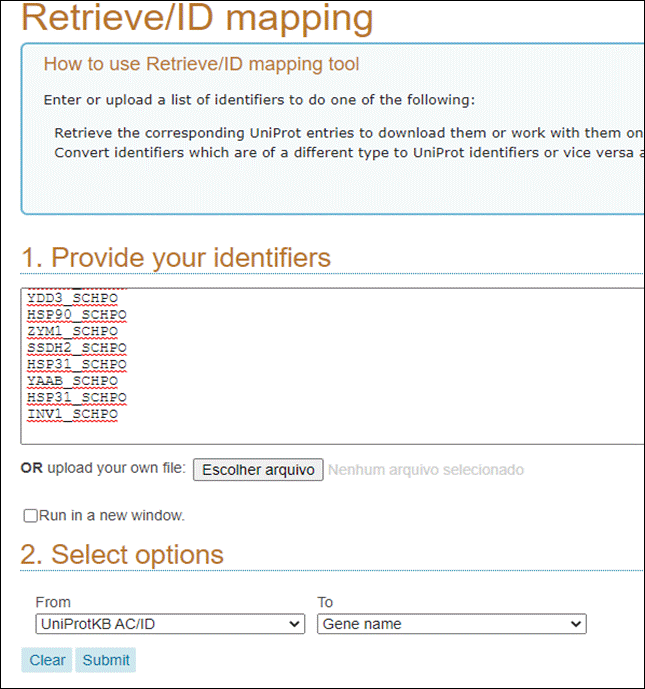
Irá abrir então uma página contendo todos os gene names dos seus identificadores:
7° Passo:
Retorne à página anterior do seu browser para fazermos uma nova busca utilizando os mesmos identificadores. Desta vez, vamos selecionar To KEGG dentro do grupo de Genome Annotation Databases:
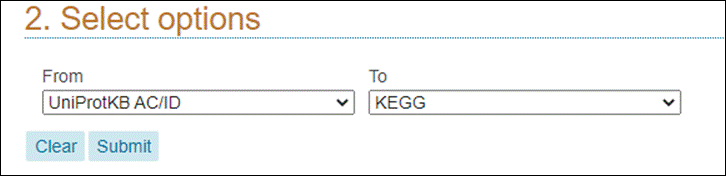
Agora irá abrir uma página contendo os identificadores KEGG que dão acesso diretamente a esse banco de dados:
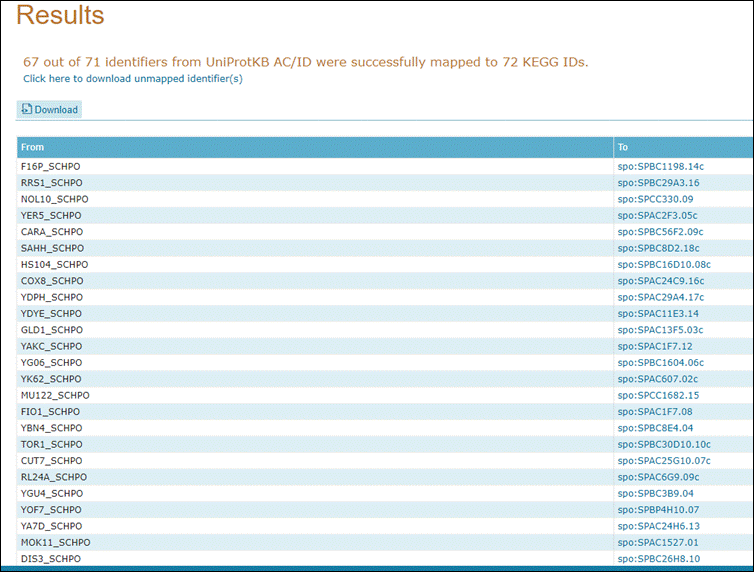
14° Passo:
Vamos explorar alguma dessas vias. Clique na primeira opção de KEGG com o botão direito e vá em Abrir em uma nova guia. Expore as proteínas: SPBC56F2.09c e SPBC8D2.18c.

Na aba que irá abrir, será do banco de dados do KEGG:
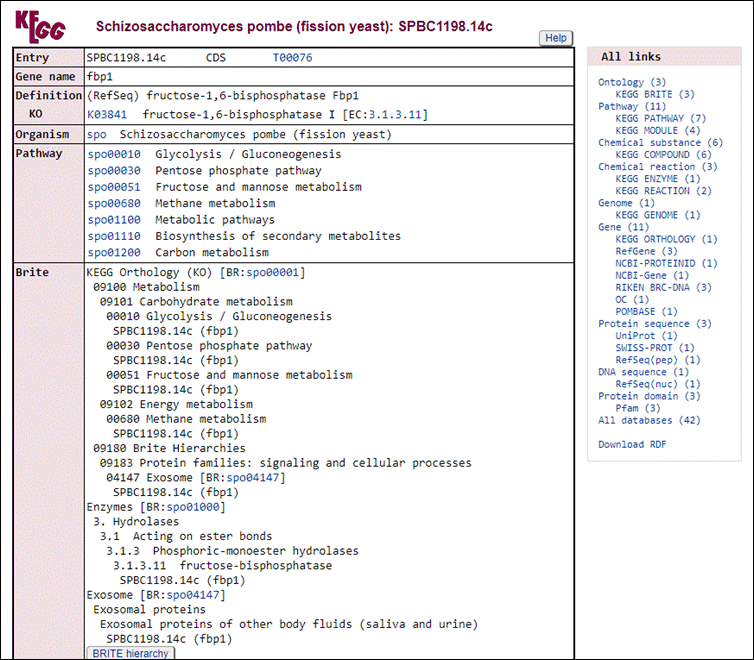
A partir daqui nós podemos visualizar todas as vias metabólicas que este gene está envolvido, como por exemplo, a spo00010 Glycolysis/Gluconeogenesis. Vamos fazer o mesmo procedimento poder abrir este link em uma nova aba do navegador:
Nesta nova aba aberta agora podemos explorar em qual região da via este gene está com sua expressão diferenciada. Neste caso, está em vermelho o número 3.1.3.11 dentro da parte de Pentose phophate pathway.
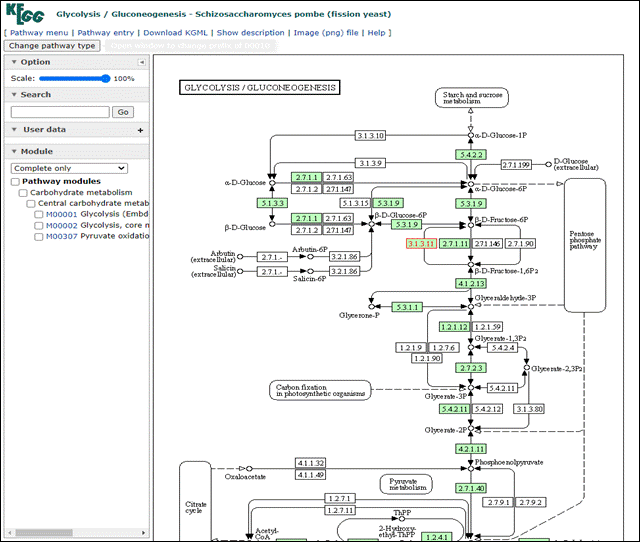
Agora você pode explorar todas as vias e genes que você identificou como diferencialmente expressos.
8° Passo:
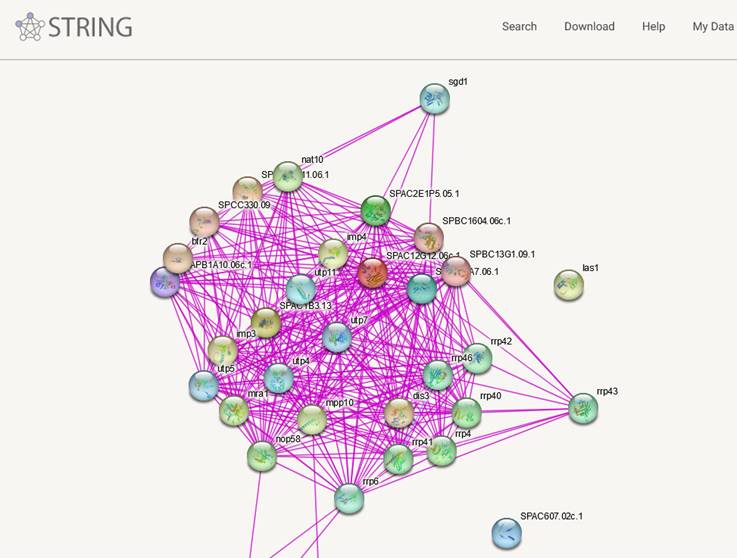
Uma outra maneira de explorar genes diferencialmente expressos é a biologia de sistemas, Entre no site do String: . https://string-db.org/ e clique em SEARCH
Opte na barra lateral por Multiple proteins
Indique o organismo Schizosaccharomyces pombe
Aceite a conferência da 91 entradas e veja a rede de interações PPI
Melhor trocar em Settings para High confidence 0.7
Peça para mostrar não mais que 10 interações com a base de dados e UPDATE
Veja em Analysis as vias mais enriquecidas
Teste o clustering com MCL e inflation 2, selecione o maior cluster
Volte em Settings e deixe só as arestas de dados experimentias
Estes são os parâmetros a explorar no STRING